Здравствуйте, гость ( Вход | Регистрация )
 12.06.2018 - 08:32 12.06.2018 - 08:32
Сообщение
#1
|
|
|
Группа: Пользователи Сообщений: 10 Регистрация: 30.04.2018 Пользователь №: 31313 |
Прикрепленные файлы
|
|
|
 |
 |
 |
Ответов
 13.06.2018 - 12:43 13.06.2018 - 12:43
Сообщение
#2
|
|
|
Группа: Пользователи Сообщений: 1091 Регистрация: 26.08.2010 Пользователь №: 22699 |
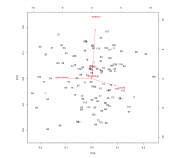
Код df.cin <- data.table::fread("./CIN.csv", dec=",") str(df.cin) df.cin$КИН <- as.factor(df.cin$КИН) df.cin$пол <- as.factor(df.cin$пол) df.cin$поч_функц <- as.factor(df.cin$поч_функц) library(mice) md.pattern(df.cin) library(randomForestSRC) ?rfsrc res.rfsrc <- rfsrc(КИН~., data=na.omit(df.cin[,-1]), case.wt = randomForestSRC:::make.wt(na.omit(df.cin)$КИН), sampsize = randomForestSRC:::make.size(na.omit(df.cin)$КИН), ntree = 5000) res.rfsrc <- rfsrc(КИН~., data=df.cin[,-1], case.wt = randomForestSRC:::make.wt(df.cin$КИН), sampsize = randomForestSRC:::make.size(df.cin$КИН), na.action = "na.impute") res.rfsrc <- rfsrc(КИН~., data=df.cin[,-1], na.action = "na.impute") res.rfsrc randomForestSRC::var.select(res.rfsrc, nrep = 40) > res.rfsrc Sample size: 116 Frequency of class labels: 100, 16 Number of trees: 5000 Forest terminal node size: 1 Average no. of terminal nodes: 8.6332 No. of variables tried at each split: 3 Total no. of variables: 9 Analysis: RF-C Family: class Splitting rule: gini Normalized Brier score: 80.79 Error rate: 0.27, 0.22, 0.56 Confusion matrix: predicted observed 0 1 class.error 0 78 22 0.2200 1 9 7 0.5625 Overall error rate: 26.72% > randomForestSRC::var.select(res.rfsrc, nrep = 40) minimal depth variable selection ... ----------------------------------------------------------- family : class var. selection : Minimal Depth conservativeness : medium x-weighting used? : TRUE dimension : 9 sample size : 116 ntree : 5000 nsplit : 0 mtry : 3 nodesize : 1 refitted forest : FALSE model size : 5 depth threshold : 2.9802 PE (true OOB) : 0.2672 0.22 0.5625 Top variables: depth vimp возраст 2.633 NA тропонин 2.783 NA КФК2 2.944 NA калий 2.950 NA креатинин 2.976 NA ----------------------------------------------------------- > Вполне себе выделяет... Правда что то разумное сказать почему выделяет как то не очень получается... ну возрас большой, калий большой, малый креатинин ??? Но какое то решающее правило не выводиться, да и случайный лес меняет показания постоянно... Код data.cin.pca <- data.frame(КИН=na.omit(df.cin[,-c(1,4,6,8,10)])[,c("КИН")],
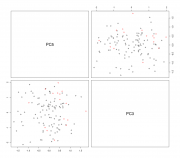
prcomp(na.omit(df.cin[,-c(1,3,4,6,8,10)]), center = T, scale. = T)$x) data.cin.pca$КИН <- as.factor(data.cin.pca$КИН) res.pca.rfsrc <- rfsrc(КИН~., data=data.cin.pca, case.wt = randomForestSRC:::make.wt(data.cin.pca$КИН), sampsize = randomForestSRC:::make.size(data.cin.pca$КИН), ntree = 5000) res.pca.rfsrc randomForestSRC::var.select(res.pca.rfsrc, nrep = 40) res.pca <- prcomp(na.omit(df.cin[,-c(1,3,4,6,8,10)]), center = T, scale. = T) biplot(res.pca, choices = c(5,3)) pairs(data.cin.pca[,c(6,4)], col = c("black", "red")[data.cin.pca$КИН]) Сообщение отредактировал p2004r - 13.06.2018 - 12:46  |
|
|
 |
 |
Сообщений в этой теме
 Felix77 Модель для прогнозирования. 12.06.2018 - 08:32
Felix77 Модель для прогнозирования. 12.06.2018 - 08:32
 passant Цитата(Felix77 @ 12.06.2018 - 08:32)... 12.06.2018 - 09:23
passant Цитата(Felix77 @ 12.06.2018 - 08:32)... 12.06.2018 - 09:23
 100$ А вот интересно, почему при выращивании леса узлы ... 13.06.2018 - 14:03
100$ А вот интересно, почему при выращивании леса узлы ... 13.06.2018 - 14:03

 p2004r Цитата(100$ @ 13.06.2018 - 14:0... 13.06.2018 - 15:34
p2004r Цитата(100$ @ 13.06.2018 - 14:0... 13.06.2018 - 15:34
 100$ Понятно.
А у randomForest'a SRC (я даже и не з... 13.06.2018 - 16:02
100$ Понятно.
А у randomForest'a SRC (я даже и не з... 13.06.2018 - 16:02
 p2004r Цитата(100$ @ 13.06.2018 - 16:0... 13.06.2018 - 19:44
p2004r Цитата(100$ @ 13.06.2018 - 16:0... 13.06.2018 - 19:44  |