Здравствуйте, гость ( Вход | Регистрация )
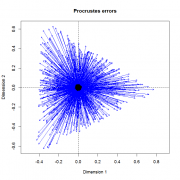
 23.11.2017 - 16:13 23.11.2017 - 16:13
Сообщение
#1
|
|
|
Группа: Пользователи Сообщений: 19 Регистрация: 15.12.2011 Пользователь №: 23369 |
Имеются данные (процентное содержание фракций A, B и C (столбцы 2-4) и общее количество (столбец 1) определенных липидов в плазме), взятые у одного контрольного индивида (1) и полсотни испытуемых (2-48) после воздействия неким агентом. Как правильно статистически обработать эти данные, и какие и чем обоснованные выводы в результате можно сделать?
Прикрепленные файлы
|
|
|
 |
 |
 |
Ответов
(1 - 22)
 23.11.2017 - 19:49 23.11.2017 - 19:49
Сообщение
#2
|
|
|
Группа: Пользователи Сообщений: 1091 Регистрация: 26.08.2010 Пользователь №: 22699 |
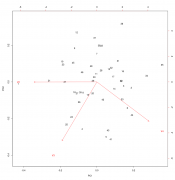
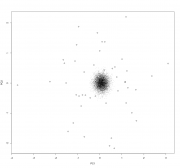
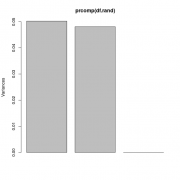
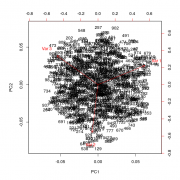
Имеются данные (процентное содержание фракций A, B и C (столбцы 2-4) и общее количество (столбец 1) определенных липидов в плазме), взятые у одного контрольного индивида (1) и полсотни испытуемых (2-48) после воздействия неким агентом. Как правильно статистически обработать эти данные, и какие и чем обоснованные выводы в результате можно сделать? "Обработать" это жаргон ничего увы конкретного не означающий. 1. Очень странный набор данных. Если у нас есть точка и выборка, то все что мы можем сделать это построить процентили распределения и посмотреть на какой из них попала именно эта точка. mvtnorm: Multivariate Normal and t Distributions https://cran.r-project.org/web/packages/mvt...s/MVT_Rnews.pdf 2. Можно просто посмотреть что там в датасете "глазами" Вот например читаем данные и смотрим на большую часть дисперсии в них. Номера точки соответствуют нумерации в файле Код > df.lipid <-read.csv2("Фракционный состав.csv", header=F) > plot(prcomp(df.lipid[,2:4]/df.lipid[,1], center=T, scale.=T)) > biplot(prcomp(df.lipid[,2:4]/df.lipid[,1], center=T, scale.=T)) Ну и например можно оценить где здесь среднее арифметическое многомерного распределения, и его квантили визуально оценить бутсрепом. Код > butstrep <- do.call(rbind,
replicate(10000, predict(df.lipid.pca, data.frame(t(colMeans((df.lipid[-1,2:4]/df.lipid[-1,1])[sample(1:(48-1), replace=T),])))), simplify=F) ) > plot(df.lipid.pca$x[,1:2]) > points(butstrep[,1:2], pch=".") Сообщение отредактировал p2004r - 23.11.2017 - 19:50  |
|
|
 |
 |
 23.11.2017 - 22:43 23.11.2017 - 22:43
Сообщение
#3
|
|
|
Группа: Пользователи Сообщений: 231 Регистрация: 27.04.2016 Пользователь №: 28223 |
Ну у вас и примерчики. То выборки из четырех элментов с необходимостью провести параметрический (!) анализ различия средних. А вот теперь выборка 1+47. Прям экстрим какой-то.
Действительно, с такой выборкой трудно что-то вразумительное предложить. Если бы меня заставили это сделать (под пытками Сообщение отредактировал passant - 23.11.2017 - 22:46 |
|
|
 |
 |
 23.11.2017 - 22:46 23.11.2017 - 22:46
Сообщение
#4
|
|
|
Группа: Пользователи Сообщений: 1219 Регистрация: 13.01.2008 Из: Челябинск Пользователь №: 4704 |
>p2004r
С обычным PCA здесь засада в виде композиционного характера данных. Где проходит граница допустимой степени "композиционности" не известно. Но традиционно для долей процентов (типа миллиграммы на литр или килограмм) ей пренебрегают и скорее всего обосновано. Но когда речь идёт о % и десятках процентов, композиции будут натягивать ложные корреляции. С 1990-х для многомерного анализа композиционных данных используют статистику Эйчисона, в т.ч. специальные предварительные преобразования "разворачивающие" constrained данные в как бы независимые. Разбирался давно и использовал ещё аддон к экселю "CoDaPack". Сейчас это есть в r, но пока не было подходящей задачи: http://www.stat.boogaart.de/compositions/ https://cran.r-project.org/web/packages/rob...ions/index.html |
|
|
 |
 |
 24.11.2017 - 01:35 24.11.2017 - 01:35
Сообщение
#5
|
|
|
Группа: Пользователи Сообщений: 1091 Регистрация: 26.08.2010 Пользователь №: 22699 |
>p2004r С обычным PCA здесь засада в виде композиционного характера данных. Где проходит граница допустимой степени "композиционности" не известно. Но традиционно для долей процентов (типа миллиграммы на литр или килограмм) ей пренебрегают и скорее всего обосновано. Но когда речь идёт о % и десятках процентов, композиции будут натягивать ложные корреляции. С 1990-х для многомерного анализа композиционных данных используют статистику Эйчисона, в т.ч. специальные предварительные преобразования "разворачивающие" constrained данные в как бы независимые. Разбирался давно и использовал ещё аддон к экселю "CoDaPack". Сейчас это есть в r, но пока не было подходящей задачи: http://www.stat.boogaart.de/compositions/ https://cran.r-project.org/web/packages/rob...ions/index.html PCA прекрасно расправляет "смеси" в тернарные трафики, отжимая лишнюю размерность. Здесь просто крайне мал размах варьирования состава, вот и не видно результирующего "треугольника(пирамиды)".  |
|
|
 |
 |
 24.11.2017 - 08:56 24.11.2017 - 08:56
Сообщение
#6
|
|
|
Группа: Пользователи Сообщений: 1091 Регистрация: 26.08.2010 Пользователь №: 22699 |
>p2004r С обычным PCA здесь засада в виде композиционного характера данных. Где проходит граница допустимой степени "композиционности" не известно. Но традиционно для долей процентов (типа миллиграммы на литр или килограмм) ей пренебрегают и скорее всего обосновано. Но когда речь идёт о % и десятках процентов, композиции будут натягивать ложные корреляции. С 1990-х для многомерного анализа композиционных данных используют статистику Эйчисона, в т.ч. специальные предварительные преобразования "разворачивающие" constrained данные в как бы независимые. Разбирался давно и использовал ещё аддон к экселю "CoDaPack". Сейчас это есть в r, но пока не было подходящей задачи: http://www.stat.boogaart.de/compositions/ https://cran.r-project.org/web/packages/rob...ions/index.html Вот иллюстрация (именно для больших колебаний состава) Код > df.rand <- t(apply(t(replicate(1000, runif(3))), 1, function(d) d/sum(d))) > str(df.rand) num [1:1000, 1:3] 0.3748 0.4819 0.0077 0.2919 0.42 ... > head(df.rand) [,1] [,2] [,3] [1,] 0.374779178 0.32692176 0.29829906 [2,] 0.481935449 0.02743644 0.49062811 [3,] 0.007700625 0.85640582 0.13589356 [4,] 0.291939338 0.62626290 0.08179776 [5,] 0.420032905 0.42936418 0.15060291 [6,] 0.497522641 0.05128767 0.45118969 > plot(prcomp(df.rand)) > biplot(prcomp(df.rand)) Для многомерных данных все также. Вот вариант для "малых колебаний состава смеси" Код > df.rand <- t(apply(t(replicate(1000, 3+runif(3))), 1, function(d) d/sum(d))) > str(df.rand) num [1:1000, 1:3] 0.327 0.329 0.34 0.335 0.321 ... > head(df.rand) [,1] [,2] [,3] [1,] 0.3271537 0.3089877 0.3638586 [2,] 0.3286260 0.3195480 0.3518260 [3,] 0.3397769 0.3595972 0.3006259 [4,] 0.3351038 0.3093977 0.3554985 [5,] 0.3206596 0.3115080 0.3678324 [6,] 0.3065733 0.3678315 0.3255952 > plot(prcomp(df.rand)) > biplot(prcomp(df.rand))  |
|
|
 |
 |
 24.11.2017 - 10:30 24.11.2017 - 10:30
Сообщение
#7
|
|
|
Группа: Пользователи Сообщений: 19 Регистрация: 15.12.2011 Пользователь №: 23369 |
2. Можно просто посмотреть что там в датасете "глазами" Визуально я вижу, что агент слегка уменьшает количество фракции А, очень сильно увеличивает количество фракции С, но не влияет на фракцию В. Общее количество также не меняется. Но вряд ли можно об этом (или о другом возможном выводе) написать без приведения каких-либо подтверждающих статистик или графиков. А здесь нужно именно написать. Код > df.lipid <-read.csv2("Фракционный состав.csv", header=F) > plot(prcomp(df.lipid[,2:4]/df.lipid[,1], center=T, scale.=T)) > biplot(prcomp(df.lipid[,2:4]/df.lipid[,1], center=T, scale.=T)) Это какая конкретно программа (и версия) и где ее взять? И как туда ввести приведенный код? я бы психанул и провел кластеризацию Этого пока мне маловато - можно конкретнее, пожалуйста, какой общедоступной программой и как именно? PS Остальные комменты в процессе... |
|
|
 |
 |
 24.11.2017 - 11:06 24.11.2017 - 11:06
Сообщение
#8
|
|
|
Группа: Пользователи Сообщений: 1091 Регистрация: 26.08.2010 Пользователь №: 22699 |
Это какая конкретно программа (и версия) и где ее взять? И как туда ввести приведенный код? https://ru.wikipedia.org/wiki/R_(%D1%8F%D0%...BD%D0%B8%D1%8F)  |
|
|
 |
 |
 24.11.2017 - 11:13 24.11.2017 - 11:13
Сообщение
#9
|
|
|
Группа: Пользователи Сообщений: 1091 Регистрация: 26.08.2010 Пользователь №: 22699 |
Визуально я вижу, что агент слегка уменьшает количество фракции А, очень сильно увеличивает количество фракции С, но не влияет на фракцию В. Настоящие влияния агента не совсем такие. А соответствуют влиянию агента и ничему больше. В соответствуют влиянию агента, но это влияние слабо + B имеет собственную динамику С соответствует влиянию агента сильнее чем A, но "в противофазе".  |
|
|
 |
 |
 24.11.2017 - 14:38 24.11.2017 - 14:38
Сообщение
#10
|
|
|
Группа: Пользователи Сообщений: 231 Регистрация: 27.04.2016 Пользователь №: 28223 |
Этого пока мне маловато - можно конкретнее, пожалуйста, какой общедоступной программой ? Ответ уже дал уважаемый p2004r - R https://www.r-project.org/ и как именно? Существуют десятки пакетов для R с реализацией различных алгоритмов кластерного анализа: https://www.rdocumentation.org/taskviews#Cluster https://cran.r-project.org/web/views/Cluster.html Выбирайте любой. Или тот, описание к которому легче найти в сети. Сообщение отредактировал passant - 24.11.2017 - 14:41 |
|
|
 |
 |
 24.11.2017 - 18:45 24.11.2017 - 18:45
Сообщение
#11
|
|
|
Группа: Пользователи Сообщений: 19 Регистрация: 15.12.2011 Пользователь №: 23369 |
Код > df.lipid <-read.csv2("Фракционный состав.csv", header=F) > plot(prcomp(df.lipid[,2:4]/df.lipid[,1], center=T, scale.=T)) > biplot(prcomp(df.lipid[,2:4]/df.lipid[,1], center=T, scale.=T)) Да, теперь это работает, спасибо. Однако, чтобы ответ был полезен не только тому, кто отвечает, но и тому, кто задал вопрос, необходимо объяснить, что показано на рисунках (наверно, это рис.1 и 2). Пока что для меня это просто какая-то дисперсия в 3 столбцах и какое-то рассеяние с какими-то векторами v2-v4 по каким-то осям pc1 и pc2. Можете подробно объяснить? Код > butstrep <- do.call(rbind, replicate(10000, predict(df.lipid.pca, data.frame(t(colMeans((df.lipid[-1,2:4]/df.lipid[-1,1])[sample(1:(48-1), replace=T),])))), simplify=F) ) > plot(df.lipid.pca$x[,1:2]) > points(butstrep[,1:2], pch=".") Это не работает (наверно это рис.3): Ошибка в predict(df.lipid.pca, data.frame(t(colMeans((df.lipid[-1, 2:4]/df.lipid[-1, : объект 'df.lipid.pca' не найден Цитата(passant) Существуют десятки пакетов для R с реализацией различных алгоритмов кластерного анализа Да, по Вашим ссылкам полно пакетов, спасибо. Но внятного объяснения с примером пока не нашлось. Поищу еще на досуге (хотя может такого объяснения и нет). |
|
|
 |
 |
 24.11.2017 - 21:55 24.11.2017 - 21:55
Сообщение
#12
|
|
|
Группа: Пользователи Сообщений: 1091 Регистрация: 26.08.2010 Пользователь №: 22699 |
Да, теперь это работает, спасибо. Однако, чтобы ответ был полезен не только тому, кто отвечает, но и тому, кто задал вопрос, необходимо объяснить, что показано на рисунках (наверно, это рис.1 и 2). Пока что для меня это просто какая-то дисперсия в 3 столбцах и какое-то рассеяние с какими-то векторами v2-v4 по каким-то осям pc1 и pc2. Можете подробно объяснить? Это не работает (наверно это рис.3): Ошибка в predict(df.lipid.pca, data.frame(t(colMeans((df.lipid[-1, 2:4]/df.lipid[-1, : объект 'df.lipid.pca' не найден Да, по Вашим ссылкам полно пакетов, спасибо. Но внятного объяснения с примером пока не нашлось. Поищу еще на досуге (хотя может такого объяснения и нет). Да, пропущена строчка сохранения результатов PCA, но сам PCA явно проводиться в первой части приведенного кода. Просто сохраните его. df.lipid.pca <- prcomp(df.lipid[,2:4]/df.lipid[,1], center=T, scale.=T)  |
|
|
 |
 |
 25.11.2017 - 20:14 25.11.2017 - 20:14
Сообщение
#13
|
|
|
Группа: Пользователи Сообщений: 1091 Регистрация: 26.08.2010 Пользователь №: 22699 |
Да, теперь это работает, спасибо. Однако, чтобы ответ был полезен не только тому, кто отвечает, но и тому, кто задал вопрос, необходимо объяснить, что показано на рисунках (наверно, это рис.1 и 2). Пока что для меня это просто какая-то дисперсия в 3 столбцах и какое-то рассеяние с какими-то векторами v2-v4 по каким-то осям pc1 и pc2. Можете подробно объяснить? Прочитайте что такое тернарный график https://en.wikipedia.org/wiki/Ternary_plot и как работать с данными о составе смесей (или результатов голосований). Собственно тернарный рафик в результате PCA датасета составов смесей и получается всегда. Можно взять пакет R http://www.ggtern.com/ и построить процентили распределения смеси липидов в нем. Сообщение отредактировал p2004r - 25.11.2017 - 20:16  |
|
|
 |
 |
 25.11.2017 - 22:48 25.11.2017 - 22:48
Сообщение
#14
|
|
|
Группа: Пользователи Сообщений: 107 Регистрация: 27.12.2015 Пользователь №: 27815 |
2 p2004r
Добрый вечер. У Вас df.rand1 <- t(apply(t(replicate(1000, runif(3))), 1, function(d) d/sum(d))) df.rand2 <- t(apply(t(replicate(1000, 3+runif(3))), 1, function(d) d/sum(d))) p1 <- prcomp(df.rand1) p2 <- prcomp(df.rand2) Можете показать пример, как "сравнить/обработать" p1 и p2 и заполучить "заветное p<0.05"? |
|
|
 |
 |
 26.11.2017 - 20:02 26.11.2017 - 20:02
Сообщение
#15
|
|
|
Группа: Пользователи Сообщений: 1091 Регистрация: 26.08.2010 Пользователь №: 22699 |
2 p2004r Добрый вечер. У Вас df.rand1 <- t(apply(t(replicate(1000, runif(3))), 1, function(d) d/sum(d))) df.rand2 <- t(apply(t(replicate(1000, 3+runif(3))), 1, function(d) d/sum(d))) p1 <- prcomp(df.rand1) p2 <- prcomp(df.rand2) Можете показать пример, как "сравнить/обработать" p1 и p2 и заполучить "заветное p<0.05"? У меня не так. У меня сделан predict() по однократно сделанному анализу. Процентили для набора смесей считает пакет из поста (где то целая книжка попадалась как в R работать с данными о процентном составе смесей, сейчас не соображу).  |
|
|
 |
 |
 27.11.2017 - 06:51 27.11.2017 - 06:51
Сообщение
#16
|
|
|
Группа: Пользователи Сообщений: 1219 Регистрация: 13.01.2008 Из: Челябинск Пользователь №: 4704 |
PCA прекрасно расправляет "смеси" в тернарные трафики, отжимая лишнюю размерность. Здесь просто крайне мал размах варьирования состава, вот и не видно результирующего "треугольника(пирамиды)". Прикольно! У меня никогда не было столько данных, чтобы увидеть треугольник, а с симуляциями не играл. ... Можете показать пример, как "сравнить/обработать" p1 и p2 и заполучить "заветное p<0.05"? Сравнить два набора PC можно прокрустовым анализом с расчётом р рандомизационным критерием. library(vegan) protest(p1, p2, scores = "sites", permutations = how(nperm = 999)) #можно посмотреть статистику и разности матриц после прокрустова преобразования для определения наиболее сильных невязок: difpc12<-procrustes(p1,p2) summary(difpc12) plot(difpc12) plot(difpc12, kind=2) Сообщение отредактировал nokh - 27.11.2017 - 07:13 |
|
|
 |
 |
 27.11.2017 - 14:51 27.11.2017 - 14:51
Сообщение
#17
|
|
|
Группа: Пользователи Сообщений: 19 Регистрация: 15.12.2011 Пользователь №: 23369 |
пропущена строчка сохранения результатов PCA df.lipid.pca <- prcomp(df.lipid[,2:4]/df.lipid[,1], center=T, scale.=T) Вот сейчас все работает, рисунки строятся, спасибо. Теперь осталось самое главное и непонятное - интерпретировать полученные картинки... больше ничего в голову и не приходит А если добавить контрольных индивидов, чтобы их было не 1, а хотя бы 2? В принципе ведь контрольных никто никаким агентом не колет, так что можно сделать анализ фракций у любого. Это как-то может помочь? Типа статист. доказать, что различия по общему и фракционному составу до и после воздействия агентом имеются (или не имеются)? Или максимум моих данных - визуально на них посмотреть? |
|
|
 |
 |
 27.11.2017 - 17:59 27.11.2017 - 17:59
Сообщение
#18
|
|
|
Группа: Пользователи Сообщений: 902 Регистрация: 23.08.2010 Пользователь №: 22694 |
|
|
|
 |
 |
 28.11.2017 - 20:27 28.11.2017 - 20:27
Сообщение
#19
|
|
|
Группа: Пользователи Сообщений: 107 Регистрация: 27.12.2015 Пользователь №: 27815 |
2 p2004r, nokh
Спасибо за информацию. Оставлю то, что нагуглил: http://www.compositionaldata.com/material.php -можно скачать скрипты, слайды, другую информацию касательно составных данных. Analyzing Compositional Data with R (Use R!) 2,013th Edition (K. Gerald van den Boogaart, Raimon Tolosana-Delgado) - книжка афтаров представенноговыше пакета, доступна на Амазоне. |
|
|
 |
 |
 29.11.2017 - 00:25 29.11.2017 - 00:25
Сообщение
#20
|
|
|
Группа: Пользователи Сообщений: 1219 Регистрация: 13.01.2008 Из: Челябинск Пользователь №: 4704 |
Я тут подумал и решил усомниться в исходно композиционном характере данных. Очень часто люди загоняют в композиции полноценные (ну или почти полноценные данные), т.к. смешивают статистический анализ с удобством интерпретации. Интерпретировать иногда, действительно, удобнее в %, но если человек пару-тройку раз прошёл через ад всяких лог-преобразований Эйчисона со структурными нулями и с потолка взятыми значениями для неструктурных нулей, то остатвит всё это на тогда, когда без этого вообще никак не обойтись.
Например, аммоний, нитриты и нитраты в воде являются последовательными стадиями окисления азота органического происхождения. Они выражаются в мг/л, т.е. строго говоря это тоже композиция, но речь идёт о миллионных долях (ppm), когда композиция никак не влияет на связь между показателями. Если для интерпретации возникает желание оперировать %-ным соотношением азота в разных формах, то пересчёт на % даст полноценную композицию со всеми вытекающими проблемами. В то же время хлориды, сульфаты и гидрокарбонаты в воде представляют собой классическую композицию: они выражаются в % от общего состава и если чего-то станет меньше, то остального станет больше автоматически - здесь придётся повозиться. Кстати, если геологи почти сразу пришли к грамотному анализу таких данных, то гидрохимики уже 25 лет в пути, медики (в массе) - в начале пути (формула крови - композиция), а биологи (в массе) даже не слышали про такое. Оно и понятно, проблемы появляются только при многомерном анализе, а до него многие просто не дорастают. >Света К Если ваши фракции исходно выражаются, скажем, в миллимолях на литр, то нужно пересчитать % в эти исходные величины и забыть про % как страшный сон. Вне жёсткой композиции можно прекрасно проверить 1 наблюдение на многомерный выброс по расстоянию Махаланобиса (с расчётом р-значения), а для 2х наблюдений - провести дискриминантный анализ (т.к. это - "совсем другое дело":)). Но опять-таки, это - чисто технические вещи, ими научную методологию не заменить. Сообщение отредактировал nokh - 29.11.2017 - 00:33 |
|
|
 |
 |
 29.11.2017 - 01:09 29.11.2017 - 01:09
Сообщение
#21
|
|
|
Группа: Пользователи Сообщений: 19 Регистрация: 15.12.2011 Пользователь №: 23369 |
читаем данные и смотрим на большую часть дисперсии в них. Номера точки соответствуют нумерации в файле Это я так понимаю второй рисунок? А на первом что за 3 разных столбца с дисперсией? Фракция А варьирует значительно сильнее чем В, а В сильнее чем С? Можете объяснить? Цитата(p2004r) Прочитайте что такое тернарный график https://en.wikipedia.org/wiki/Ternary_plot Читаю данное пояснение и вижу, что это все-таки не то, даже совсем не то. Тут написано, что тернарные графики используются для показа состава смесей, состоящих из 3-х компонентов или показа условий когда три отдельные фазы существуют в равновесии. У меня ничего такого нет, да я и так знаю, что эти фракции у каждого индивида существуют в равновесии, у меня есть действующий агент, и надо бы статистически доказать повлиял/не повлиял ли он (и как именно) на общий/фракционный состав смеси. Т.е. есть ли какой-либо эффект он него? Да и не 3 компонента может быть, а до 10 (хотя какая разница), сейчас например я делаю опыт где определяю уже 5 фракций. Цитата(passant) Существуют десятки пакетов для R с реализацией различных алгоритмов кластерного анализа...Выбирайте любой. Что-то не получается. Беру первый попавшийся пакет cluster с настройками по умолчанию - строит дендрограмму, но разделения на отдельные группы там нет. Есть в пакете еще куча всяких опций, но разобраться в них самостоятельно я не смогу. |
|
|
 |
 |
 29.11.2017 - 12:25 29.11.2017 - 12:25
Сообщение
#22
|
|
|
Группа: Пользователи Сообщений: 1091 Регистрация: 26.08.2010 Пользователь №: 22699 |
Это я так понимаю второй рисунок? А на первом что за 3 разных столбца с дисперсией? Фракция А варьирует значительно сильнее чем В, а В сильнее чем С? Можете объяснить? Читаю данное пояснение и вижу, что это все-таки не то, даже совсем не то. Тут написано, что тернарные графики используются для показа состава смесей, состоящих из 3-х компонентов или показа условий когда три отдельные фазы существуют в равновесии. У меня ничего такого нет, да я и так знаю, что эти фракции у каждого индивида существуют в равновесии, у меня есть действующий агент, и надо бы статистически доказать повлиял/не повлиял ли он (и как именно) на общий/фракционный состав смеси. Т.е. есть ли какой-либо эффект он него? Да и не 3 компонента может быть, а до 10 (хотя какая разница), сейчас например я делаю опыт где определяю уже 5 фракций. 1) Прочитайте сначала https://ru.wikipedia.org/wiki/%D0%9C%D0%B5%...%B5%D0%BD%D1%82 потом будет уместно задавать вопросы. 2) я поделил на количество, это видите? (хотя, какая разница)  |
|
|
 |
 |
 1.12.2017 - 15:47 1.12.2017 - 15:47
Сообщение
#23
|
|
|
Группа: Пользователи Сообщений: 19 Регистрация: 15.12.2011 Пользователь №: 23369 |
Ответить раньше не было возможности, - другие идеи, другой эксперимент, другие анализы...
1) Прочитайте сначала ... Спасибо, но пока не понятно. По вашей ссылке написано, что на рис.2 проведен тест на уменьшение размерности данных трех моих фракций (хотя какая разница сколько фракций), а красные линии - это линии вдоль которых дисперсия по каждой фракции максимальна. Т.е. Вы вроде бы свели всю матрицу моих данных по 3 фракциям подопытных к трем векторам и возможно варьирование фракции А - это линия V2, а фракции С - это V4. Если это так - то тут понятно. Ну а дальше что? Что обозначают оси, причем разные, с разными обозначениями, и с отрицательными значениями, куда направлены вектора, и, самое главное, как это описывать и подавать; что важно, что - нет. Мне кажется, что Вы просто "забываете", что я не знаю этого вопроса, поэтому нуждаюсь в конкретных ответах, а не в пересылках на другие ресурсы (хотя, наверняка полезных). Цитата(p2004r) я поделил на количество, это видите? Нет, из такого объяснения не видно что на что Вы делили. Может Вы конечно объясняете не мне, а самому себе (хотя, возможно, какая вам разница), тогда, наверное, и видно. Но если вы все-таки хотите помочь разобраться именно мне, тогда что показывает и как интерпретировать каждый из 3-х столбцов с дисперсией на рис.1 - пока не видно. |
|
|
 |
 |
  |