Здравствуйте, гость ( Вход | Регистрация )
 23.11.2017 - 16:13 23.11.2017 - 16:13
Сообщение
#1
|
|
|
Группа: Пользователи Сообщений: 19 Регистрация: 15.12.2011 Пользователь №: 23369 |
Имеются данные (процентное содержание фракций A, B и C (столбцы 2-4) и общее количество (столбец 1) определенных липидов в плазме), взятые у одного контрольного индивида (1) и полсотни испытуемых (2-48) после воздействия неким агентом. Как правильно статистически обработать эти данные, и какие и чем обоснованные выводы в результате можно сделать?
Прикрепленные файлы
|
|
|
 |
 |
 |
Ответов
 23.11.2017 - 22:46 23.11.2017 - 22:46
Сообщение
#2
|
|
|
Группа: Пользователи Сообщений: 1219 Регистрация: 13.01.2008 Из: Челябинск Пользователь №: 4704 |
>p2004r
С обычным PCA здесь засада в виде композиционного характера данных. Где проходит граница допустимой степени "композиционности" не известно. Но традиционно для долей процентов (типа миллиграммы на литр или килограмм) ей пренебрегают и скорее всего обосновано. Но когда речь идёт о % и десятках процентов, композиции будут натягивать ложные корреляции. С 1990-х для многомерного анализа композиционных данных используют статистику Эйчисона, в т.ч. специальные предварительные преобразования "разворачивающие" constrained данные в как бы независимые. Разбирался давно и использовал ещё аддон к экселю "CoDaPack". Сейчас это есть в r, но пока не было подходящей задачи: http://www.stat.boogaart.de/compositions/ https://cran.r-project.org/web/packages/rob...ions/index.html |
|
|
 |
 |
 24.11.2017 - 08:56 24.11.2017 - 08:56
Сообщение
#3
|
|
|
Группа: Пользователи Сообщений: 1091 Регистрация: 26.08.2010 Пользователь №: 22699 |
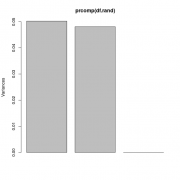
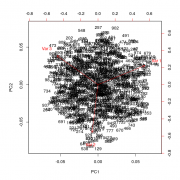
>p2004r С обычным PCA здесь засада в виде композиционного характера данных. Где проходит граница допустимой степени "композиционности" не известно. Но традиционно для долей процентов (типа миллиграммы на литр или килограмм) ей пренебрегают и скорее всего обосновано. Но когда речь идёт о % и десятках процентов, композиции будут натягивать ложные корреляции. С 1990-х для многомерного анализа композиционных данных используют статистику Эйчисона, в т.ч. специальные предварительные преобразования "разворачивающие" constrained данные в как бы независимые. Разбирался давно и использовал ещё аддон к экселю "CoDaPack". Сейчас это есть в r, но пока не было подходящей задачи: http://www.stat.boogaart.de/compositions/ https://cran.r-project.org/web/packages/rob...ions/index.html Вот иллюстрация (именно для больших колебаний состава) Код > df.rand <- t(apply(t(replicate(1000, runif(3))), 1, function(d) d/sum(d))) > str(df.rand) num [1:1000, 1:3] 0.3748 0.4819 0.0077 0.2919 0.42 ... > head(df.rand) [,1] [,2] [,3] [1,] 0.374779178 0.32692176 0.29829906 [2,] 0.481935449 0.02743644 0.49062811 [3,] 0.007700625 0.85640582 0.13589356 [4,] 0.291939338 0.62626290 0.08179776 [5,] 0.420032905 0.42936418 0.15060291 [6,] 0.497522641 0.05128767 0.45118969 > plot(prcomp(df.rand)) > biplot(prcomp(df.rand)) Для многомерных данных все также. Вот вариант для "малых колебаний состава смеси" Код > df.rand <- t(apply(t(replicate(1000, 3+runif(3))), 1, function(d) d/sum(d))) > str(df.rand) num [1:1000, 1:3] 0.327 0.329 0.34 0.335 0.321 ... > head(df.rand) [,1] [,2] [,3] [1,] 0.3271537 0.3089877 0.3638586 [2,] 0.3286260 0.3195480 0.3518260 [3,] 0.3397769 0.3595972 0.3006259 [4,] 0.3351038 0.3093977 0.3554985 [5,] 0.3206596 0.3115080 0.3678324 [6,] 0.3065733 0.3678315 0.3255952 > plot(prcomp(df.rand)) > biplot(prcomp(df.rand))  |
|
|
 |
 |
Сообщений в этой теме
 Света K Процентное содержание фракций 23.11.2017 - 16:13
Света K Процентное содержание фракций 23.11.2017 - 16:13
 p2004r Цитата(Света K @ 23.11.2017 - 16:13)... 23.11.2017 - 19:49
p2004r Цитата(Света K @ 23.11.2017 - 16:13)... 23.11.2017 - 19:49

 Света K Цитата(p2004r @ 23.11.2017 - 20:49) ... 29.11.2017 - 01:09
Света K Цитата(p2004r @ 23.11.2017 - 20:49) ... 29.11.2017 - 01:09

 p2004r Цитата(Света K @ 29.11.2017 - 01:09)... 29.11.2017 - 12:25
p2004r Цитата(Света K @ 29.11.2017 - 01:09)... 29.11.2017 - 12:25

 Света K Ответить раньше не было возможности, - другие идеи... 1.12.2017 - 15:47
Света K Ответить раньше не было возможности, - другие идеи... 1.12.2017 - 15:47
 passant Ну у вас и примерчики. То выборки из четырех элмен... 23.11.2017 - 22:43
passant Ну у вас и примерчики. То выборки из четырех элмен... 23.11.2017 - 22:43

 p2004r Цитата(nokh @ 23.11.2017 - 22:46) ... 24.11.2017 - 01:35
p2004r Цитата(nokh @ 23.11.2017 - 22:46) ... 24.11.2017 - 01:35


 nokh Цитата(p2004r @ 24.11.2017 - 03:35) ... 27.11.2017 - 06:51
nokh Цитата(p2004r @ 24.11.2017 - 03:35) ... 27.11.2017 - 06:51
 Света K Цитата(p2004r @ 23.11.2017 - 20:49) ... 24.11.2017 - 10:30
Света K Цитата(p2004r @ 23.11.2017 - 20:49) ... 24.11.2017 - 10:30

 p2004r Цитата(Света K @ 24.11.2017 - 10:30)... 24.11.2017 - 11:06
p2004r Цитата(Света K @ 24.11.2017 - 10:30)... 24.11.2017 - 11:06

 p2004r Цитата(Света K @ 24.11.2017 - 10:30)... 24.11.2017 - 11:13
p2004r Цитата(Света K @ 24.11.2017 - 10:30)... 24.11.2017 - 11:13

 passant Цитата(Света K @ 24.11.2017 - 10:30)... 24.11.2017 - 14:38
passant Цитата(Света K @ 24.11.2017 - 10:30)... 24.11.2017 - 14:38
 Света K Цитата(p2004r @ 23.11.2017 - 20:49) ... 24.11.2017 - 18:45
Света K Цитата(p2004r @ 23.11.2017 - 20:49) ... 24.11.2017 - 18:45

 p2004r Цитата(Света K @ 24.11.2017 - 18:45)... 24.11.2017 - 21:55
p2004r Цитата(Света K @ 24.11.2017 - 18:45)... 24.11.2017 - 21:55


 Света K Цитата(p2004r @ 24.11.2017 - 22:55) ... 27.11.2017 - 14:51
Света K Цитата(p2004r @ 24.11.2017 - 22:55) ... 27.11.2017 - 14:51


 100$ Цитата(Света K @ 27.11.2017 - 14:51)... 27.11.2017 - 17:59
100$ Цитата(Света K @ 27.11.2017 - 14:51)... 27.11.2017 - 17:59

 p2004r Цитата(Света K @ 24.11.2017 - 18:45)... 25.11.2017 - 20:14
p2004r Цитата(Света K @ 24.11.2017 - 18:45)... 25.11.2017 - 20:14
 comisora 2 p2004r
Добрый вечер. У Вас
df.rand1 <- t(app... 25.11.2017 - 22:48
comisora 2 p2004r
Добрый вечер. У Вас
df.rand1 <- t(app... 25.11.2017 - 22:48

 p2004r Цитата(comisora @ 25.11.2017 - 22:48... 26.11.2017 - 20:02
p2004r Цитата(comisora @ 25.11.2017 - 22:48... 26.11.2017 - 20:02
 comisora 2 p2004r, nokh
Спасибо за информацию.
Оставлю то,... 28.11.2017 - 20:27
comisora 2 p2004r, nokh
Спасибо за информацию.
Оставлю то,... 28.11.2017 - 20:27
 nokh Я тут подумал и решил усомниться в исходно компози... 29.11.2017 - 00:25
nokh Я тут подумал и решил усомниться в исходно компози... 29.11.2017 - 00:25  |