Здравствуйте, гость ( Вход | Регистрация )
 23.11.2017 - 16:13 23.11.2017 - 16:13
Сообщение
#1
|
|
|
Группа: Пользователи Сообщений: 19 Регистрация: 15.12.2011 Пользователь №: 23369 |
Имеются данные (процентное содержание фракций A, B и C (столбцы 2-4) и общее количество (столбец 1) определенных липидов в плазме), взятые у одного контрольного индивида (1) и полсотни испытуемых (2-48) после воздействия неким агентом. Как правильно статистически обработать эти данные, и какие и чем обоснованные выводы в результате можно сделать?
Прикрепленные файлы
|
|
|
 |
 |
 |
Ответов
 23.11.2017 - 19:49 23.11.2017 - 19:49
Сообщение
#2
|
|
|
Группа: Пользователи Сообщений: 1091 Регистрация: 26.08.2010 Пользователь №: 22699 |
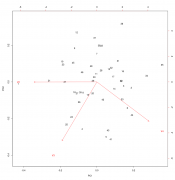
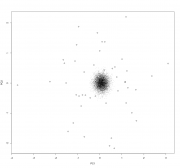
Имеются данные (процентное содержание фракций A, B и C (столбцы 2-4) и общее количество (столбец 1) определенных липидов в плазме), взятые у одного контрольного индивида (1) и полсотни испытуемых (2-48) после воздействия неким агентом. Как правильно статистически обработать эти данные, и какие и чем обоснованные выводы в результате можно сделать? "Обработать" это жаргон ничего увы конкретного не означающий. 1. Очень странный набор данных. Если у нас есть точка и выборка, то все что мы можем сделать это построить процентили распределения и посмотреть на какой из них попала именно эта точка. mvtnorm: Multivariate Normal and t Distributions https://cran.r-project.org/web/packages/mvt...s/MVT_Rnews.pdf 2. Можно просто посмотреть что там в датасете "глазами" Вот например читаем данные и смотрим на большую часть дисперсии в них. Номера точки соответствуют нумерации в файле Код > df.lipid <-read.csv2("Фракционный состав.csv", header=F) > plot(prcomp(df.lipid[,2:4]/df.lipid[,1], center=T, scale.=T)) > biplot(prcomp(df.lipid[,2:4]/df.lipid[,1], center=T, scale.=T)) Ну и например можно оценить где здесь среднее арифметическое многомерного распределения, и его квантили визуально оценить бутсрепом. Код > butstrep <- do.call(rbind,
replicate(10000, predict(df.lipid.pca, data.frame(t(colMeans((df.lipid[-1,2:4]/df.lipid[-1,1])[sample(1:(48-1), replace=T),])))), simplify=F) ) > plot(df.lipid.pca$x[,1:2]) > points(butstrep[,1:2], pch=".") Сообщение отредактировал p2004r - 23.11.2017 - 19:50  |
|
|
 |
 |
 29.11.2017 - 01:09 29.11.2017 - 01:09
Сообщение
#3
|
|
|
Группа: Пользователи Сообщений: 19 Регистрация: 15.12.2011 Пользователь №: 23369 |
читаем данные и смотрим на большую часть дисперсии в них. Номера точки соответствуют нумерации в файле Это я так понимаю второй рисунок? А на первом что за 3 разных столбца с дисперсией? Фракция А варьирует значительно сильнее чем В, а В сильнее чем С? Можете объяснить? Цитата(p2004r) Прочитайте что такое тернарный график https://en.wikipedia.org/wiki/Ternary_plot Читаю данное пояснение и вижу, что это все-таки не то, даже совсем не то. Тут написано, что тернарные графики используются для показа состава смесей, состоящих из 3-х компонентов или показа условий когда три отдельные фазы существуют в равновесии. У меня ничего такого нет, да я и так знаю, что эти фракции у каждого индивида существуют в равновесии, у меня есть действующий агент, и надо бы статистически доказать повлиял/не повлиял ли он (и как именно) на общий/фракционный состав смеси. Т.е. есть ли какой-либо эффект он него? Да и не 3 компонента может быть, а до 10 (хотя какая разница), сейчас например я делаю опыт где определяю уже 5 фракций. Цитата(passant) Существуют десятки пакетов для R с реализацией различных алгоритмов кластерного анализа...Выбирайте любой. Что-то не получается. Беру первый попавшийся пакет cluster с настройками по умолчанию - строит дендрограмму, но разделения на отдельные группы там нет. Есть в пакете еще куча всяких опций, но разобраться в них самостоятельно я не смогу. |
|
|
 |
 |
Сообщений в этой теме
 Света K Процентное содержание фракций 23.11.2017 - 16:13
Света K Процентное содержание фракций 23.11.2017 - 16:13

 p2004r Цитата(Света K @ 29.11.2017 - 01:09)... 29.11.2017 - 12:25
p2004r Цитата(Света K @ 29.11.2017 - 01:09)... 29.11.2017 - 12:25

 Света K Ответить раньше не было возможности, - другие идеи... 1.12.2017 - 15:47
Света K Ответить раньше не было возможности, - другие идеи... 1.12.2017 - 15:47
 passant Ну у вас и примерчики. То выборки из четырех элмен... 23.11.2017 - 22:43
passant Ну у вас и примерчики. То выборки из четырех элмен... 23.11.2017 - 22:43
 nokh >p2004r
С обычным PCA здесь засада в виде компо... 23.11.2017 - 22:46
nokh >p2004r
С обычным PCA здесь засада в виде компо... 23.11.2017 - 22:46

 p2004r Цитата(nokh @ 23.11.2017 - 22:46) ... 24.11.2017 - 01:35
p2004r Цитата(nokh @ 23.11.2017 - 22:46) ... 24.11.2017 - 01:35


 nokh Цитата(p2004r @ 24.11.2017 - 03:35) ... 27.11.2017 - 06:51
nokh Цитата(p2004r @ 24.11.2017 - 03:35) ... 27.11.2017 - 06:51

 p2004r Цитата(nokh @ 23.11.2017 - 22:46) ... 24.11.2017 - 08:56
p2004r Цитата(nokh @ 23.11.2017 - 22:46) ... 24.11.2017 - 08:56
 Света K Цитата(p2004r @ 23.11.2017 - 20:49) ... 24.11.2017 - 10:30
Света K Цитата(p2004r @ 23.11.2017 - 20:49) ... 24.11.2017 - 10:30

 p2004r Цитата(Света K @ 24.11.2017 - 10:30)... 24.11.2017 - 11:06
p2004r Цитата(Света K @ 24.11.2017 - 10:30)... 24.11.2017 - 11:06

 p2004r Цитата(Света K @ 24.11.2017 - 10:30)... 24.11.2017 - 11:13
p2004r Цитата(Света K @ 24.11.2017 - 10:30)... 24.11.2017 - 11:13

 passant Цитата(Света K @ 24.11.2017 - 10:30)... 24.11.2017 - 14:38
passant Цитата(Света K @ 24.11.2017 - 10:30)... 24.11.2017 - 14:38
 Света K Цитата(p2004r @ 23.11.2017 - 20:49) ... 24.11.2017 - 18:45
Света K Цитата(p2004r @ 23.11.2017 - 20:49) ... 24.11.2017 - 18:45

 p2004r Цитата(Света K @ 24.11.2017 - 18:45)... 24.11.2017 - 21:55
p2004r Цитата(Света K @ 24.11.2017 - 18:45)... 24.11.2017 - 21:55


 Света K Цитата(p2004r @ 24.11.2017 - 22:55) ... 27.11.2017 - 14:51
Света K Цитата(p2004r @ 24.11.2017 - 22:55) ... 27.11.2017 - 14:51


 100$ Цитата(Света K @ 27.11.2017 - 14:51)... 27.11.2017 - 17:59
100$ Цитата(Света K @ 27.11.2017 - 14:51)... 27.11.2017 - 17:59

 p2004r Цитата(Света K @ 24.11.2017 - 18:45)... 25.11.2017 - 20:14
p2004r Цитата(Света K @ 24.11.2017 - 18:45)... 25.11.2017 - 20:14
 comisora 2 p2004r
Добрый вечер. У Вас
df.rand1 <- t(app... 25.11.2017 - 22:48
comisora 2 p2004r
Добрый вечер. У Вас
df.rand1 <- t(app... 25.11.2017 - 22:48

 p2004r Цитата(comisora @ 25.11.2017 - 22:48... 26.11.2017 - 20:02
p2004r Цитата(comisora @ 25.11.2017 - 22:48... 26.11.2017 - 20:02
 comisora 2 p2004r, nokh
Спасибо за информацию.
Оставлю то,... 28.11.2017 - 20:27
comisora 2 p2004r, nokh
Спасибо за информацию.
Оставлю то,... 28.11.2017 - 20:27
 nokh Я тут подумал и решил усомниться в исходно компози... 29.11.2017 - 00:25
nokh Я тут подумал и решил усомниться в исходно компози... 29.11.2017 - 00:25  |