Здравствуйте, гость ( Вход | Регистрация )
 21.09.2024 - 15:02 21.09.2024 - 15:02
Сообщение
#1
|
|
|
Группа: Пользователи Сообщений: 27 Регистрация: 7.12.2012 Пользователь №: 24440 |
Товарищи, подсобите с анализом данных.
3 группы мышей: контроль, больные, леченные. Измерялось пройденное расстояние. Мыши с тяжёлыми двигательными нарушениями, поэтому по сравнению с контролем у них пройденное расстояние отличается на порядок. ANOVA и Tukey апостериорный показывают значимые отличия от контроля. но не между больными и леченными, хотя объективно между ними x2 разница. Интуиция подсказывает, что с такой разницей между средними сравнивать ANOVA некорректно. Но чем тогда и как это объяснить грамотно и какой критерий использовать? Если Стьюдента попарно использовать - выходит норм.
Прикрепленные файлы
|
|
|
 |
 |
 |
Ответов
 22.09.2024 - 22:28 22.09.2024 - 22:28
Сообщение
#2
|
|
|
Группа: Пользователи Сообщений: 107 Регистрация: 27.12.2015 Пользователь №: 27815 |
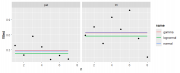
Товарищи, подсобите с анализом данных. А почему бы не рассмотреть зависимую переменную как неотрицательную величину, пик которой будет ближе к нулю, а хвосты распределения будут говорить о самых дохлых и самых выносливых мышках? Взял три модели: нормальное распределение, логнормальное и гамма. Результаты в листинге. Отношение между контролем и лечением около 2.0-2.5 (1/0.45 - 1/0.41). Много это или мало - решать Вам, но в сравнении со здоровыми это всё очень бледно смотрится. CODE library(tidyverse) library(gamlss) dat <- readxl::read_excel('YandexDisk/Data1.xlsx') |> mutate(across(where(is.character), ~as.factor(.))) |> as.data.frame() fit1 <- gamlss(value~group, data = dat, family = NO()) fit2 <- gamlss(value~group, data = dat, family = LOGNO2()) fit3 <- gamlss(value~group, data = dat, family = GAF()) df.pred <- data |> mutate( normal = predict(fit1, type = 'response', newdata = dat), lognormal = predict(fit2, type = 'response', newdata = dat), gamma = predict(fit3, type = 'response', newdata = dat) ) perf <- performance::compare_performance(fit1,fit2,fit3) pl <- df.pred |> pivot_longer(cols = c(normal, lognormal, gamma), values_to = 'fitted') |> ggplot() + geom_line(aes(x = n, y = fitted, color = name), linewidth = 1, alpha = 0.75) + geom_point(aes(x = n, y = value)) + facet_grid(name~group) posthoc1 <- pairs(emmeans::emmeans(fit1, ~group)) posthoc2 <- pairs(emmeans::emmeans(fit2, ~group, type = 'response')) posthoc3 <- pairs(emmeans::emmeans(fit3, ~group, type = 'response')) perf Name | Model | AIC (weights) | AICc (weights) | BIC (weights)| Sigma ------------------------------------------------------------------------------ fit1 | gamlss | 58.3 (<.001) | 60.5 (<.001) | 62.9 (<.001) | 1.627 fit2 | gamlss | 42.2 (0.003) | 44.4 (0.006) | 46.8 (0.006) | 1.342 fit3 | gamlss | 30.7 (0.997) | 34.3 (0.994) | 36.4 (0.994) | 1.073 posthoc1 contrast estimate SE df t.ratio p.value hc - pat 7.738 0.374 19 20.695 <.0001 hc - trt 7.362 0.361 19 20.382 <.0001 pat - trt -0.375 0.374 19 -1.004 0.5834 P value adjustment: tukey method for comparing a family of 3 estimates posthoc2 contrast ratio SE df null z.ratio p.value hc / pat 33.680 8.1281 Inf 1 14.573 <.0001 hc / trt 13.782 3.2133 Inf 1 11.252 <.0001 pat / trt 0.409 0.0988 Inf 1 -3.702 0.0006 P value adjustment: tukey method for comparing a family of 3 estimates Tests are performed on the log scale posthoc3 contrast ratio SE df null z.ratio p.value hc / pat 27.87 6.648 Inf 1 13.948 <.0001 hc / trt 12.55 2.025 Inf 1 15.677 <.0001 pat / trt 0.45 0.123 Inf 1 -2.931 0.0095 P value adjustment: tukey method for comparing a family of 3 estimates Tests are performed on the log scale
Прикрепленные файлы
|
|
|
 |
 |
Сообщений в этой теме
 Vitek_22 Сравнение трёх групп с большой разницей средних 21.09.2024 - 15:02
Vitek_22 Сравнение трёх групп с большой разницей средних 21.09.2024 - 15:02
 ИНО Большая разница средник помехой быть не может, а т... 22.09.2024 - 11:35
ИНО Большая разница средник помехой быть не может, а т... 22.09.2024 - 11:35
 nokh Как уже написал ИНО, требования модели дисперсионн... 24.09.2024 - 03:39
nokh Как уже написал ИНО, требования модели дисперсионн... 24.09.2024 - 03:39

 ИНО Цитата(nokh @ 24.09.2024 - 03:39) Дл... 24.09.2024 - 12:13
ИНО Цитата(nokh @ 24.09.2024 - 03:39) Дл... 24.09.2024 - 12:13
 Vitek_22 Спасибо за такое развёрнутое объяснение.
Да... пр... 24.09.2024 - 22:23
Vitek_22 Спасибо за такое развёрнутое объяснение.
Да... пр... 24.09.2024 - 22:23
 ИНО Простые преподаватели бессильны сколь-нибудь сущес... 25.09.2024 - 01:11
ИНО Простые преподаватели бессильны сколь-нибудь сущес... 25.09.2024 - 01:11

 Игорь Цитата(nokh @ 24.09.2024 - 04:39) Ко... 25.09.2024 - 10:29
Игорь Цитата(nokh @ 24.09.2024 - 04:39) Ко... 25.09.2024 - 10:29
 ИНО То уже другая программа, там целая куча документов... 25.09.2024 - 16:44
ИНО То уже другая программа, там целая куча документов... 25.09.2024 - 16:44
 Vitek_22 Цитата(ИНО @ 22.09.2024 - 11:35) Бол... 6.02.2025 - 19:28
Vitek_22 Цитата(ИНО @ 22.09.2024 - 11:35) Бол... 6.02.2025 - 19:28

 nokh Цитата(Vitek_22 @ 6.02.2025 - 21:28)... 9.02.2025 - 09:11
nokh Цитата(Vitek_22 @ 6.02.2025 - 21:28)... 9.02.2025 - 09:11

 Vitek_22 Цитата(nokh @ 9.02.2025 - 09:11) Как... 9.02.2025 - 22:02
Vitek_22 Цитата(nokh @ 9.02.2025 - 09:11) Как... 9.02.2025 - 22:02

 Игорь Цитата(Vitek_22 @ 9.02.2025 - 22:02)... 27.02.2025 - 11:15
Игорь Цитата(Vitek_22 @ 9.02.2025 - 22:02)... 27.02.2025 - 11:15

 ИНО Цитата(Игорь @ 27.02.2025 - 11:15) Г... 2.03.2025 - 18:17
ИНО Цитата(Игорь @ 27.02.2025 - 11:15) Г... 2.03.2025 - 18:17


 Игорь Цитата(ИНО @ 2.03.2025 - 18:17) По к... 6.03.2025 - 07:34
Игорь Цитата(ИНО @ 2.03.2025 - 18:17) По к... 6.03.2025 - 07:34

 Vitek_22 Цитата(Игорь @ 27.02.2025 - 11:15) Н... 9.04.2025 - 16:46
Vitek_22 Цитата(Игорь @ 27.02.2025 - 11:15) Н... 9.04.2025 - 16:46
 ИНО Ну, если даже после консервативнейшей в мире попра... 7.02.2025 - 18:36
ИНО Ну, если даже после консервативнейшей в мире попра... 7.02.2025 - 18:36
 ИНО Ну, если ориентироваться в выборе статистических м... 10.02.2025 - 01:00
ИНО Ну, если ориентироваться в выборе статистических м... 10.02.2025 - 01:00
 ИНО Не знаю, что там принято в доказательной медицине ... 7.03.2025 - 19:32
ИНО Не знаю, что там принято в доказательной медицине ... 7.03.2025 - 19:32  |