1. При описании количественных данных рекомендуется использовать среднее и стандартное отклонение при нормальном распределении, медиану и интерквартильный размах при ненормальном распределении. Что лучше указывать, если часть данных имеет ненормальное распределение, а часть данных нормальное? Особенно при публикации статьи, где объем ограничен. Логика мне подсказывает, что если больше переменных с ненормальным распределением, то использовать для всех медиану и интерквартильный размах. А если большая часть переменных имеет нормальное распределение, но есть несколько с ненормальным?
2. К примеру в исследовании было 100 пациентов. Исследователь решил разделить выборку на две группы (40 и 60 пациентов) в зависимости от какого-то признака. При сравнении групп различия не были выявлены. Тогда исследователь решил разделить эту же выборку по другому параметру на другие группы (30 и 70 пациентов). Насколько корректно это делать? Ведь таким образом исследователь может поступать неограниченное количество раз. Насколько корректно представлять при публикации такой подход, например в статье? Или существуют какие-то поправки?
3. Чисто практический вопрос по R. Пытаясь нарисовать график в ggplot2, столкнулся с непонятной проблемой.
Была у меня таблица, где всего два вектора: гистология (hist) и дата (date), (третий появился позже).
# A tibble: 1,580 × 3
hist date histo
<8322456> <dbl> <fctr>
1 brest 2000 brest
2 brest 2000 brest
3 brest 2001 brest
4 brest 2001 brest
5 brest 2001 brest
6 brest 2003 brest
7 brest 2003 brest
8 brest 2003 brest
9 brest 2003 brest
10 brest 2003 brest
# ... with 1,570 more rows
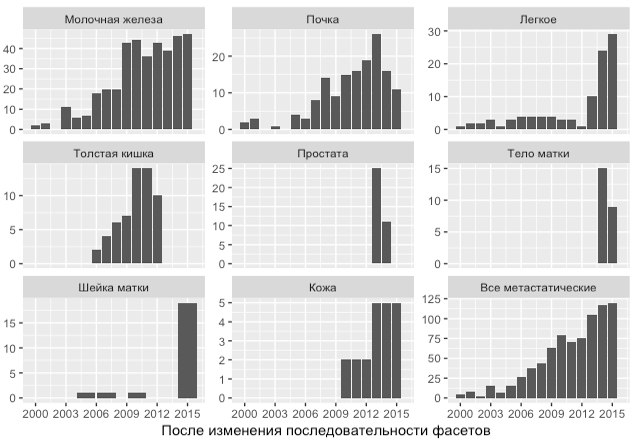
Хотел наглядно показать сколько случаев конкретной опухоли было в конкретный год. Нарисовал соответствующий график с разбивкой на фасеты, т.к. каких-то опухолей было очень много, а какие-то встречаются совсем редко.
histology <- list(
'brest'="Молочная железа",
'cerv'="Шейка матки",
'colon'="Толстая кишка",
'lung'="Легкое",
'melanoma'="Кожа",
'prost'="Простата",
'rcc'="Почка",
'uter'="Тело матки",
'metastatic'="Все метастатические")
h_labeller <- function(variable,value){
return(histology[value])}
ggplot(th, aes(x=th$date)) + geom_bar() + facet_wrap(~ th$hist, ncol=3, labeller=h_labeller, scales="free_y") +
xlab("") + ylab("") + scale_x_continuous(breaks=seq(2000, 2015, 3))
Но на последнем этапе изменения порядка фасетов почему-то теряются наблюдения на самом графике. Он становится другой (картинки прикрепил).
th$histo = factor(th$hist,
levels=c('brest','rcc','lung', 'colon', 'prost', 'uter', 'cerv', 'melanoma', 'metastatic'))
ggplot(th, aes(x=th$date)) + geom_bar() + facet_wrap(~ th$histo, ncol=3, scales="free_y") +
xlab("") + ylab("") + scale_x_continuous(breaks=seq(2000, 2015, 3))
Пожалуйста,
Уже знаю, что можно сторонними пакетами сделать фасеты из обычных графиков, но данный затык не дает покоя.